昆明动物所对树鼩KLF基因家族全长及锌指结构进行系统分析
近日,中国科学院昆明动物研究所肿瘤生物学学科组将全部17种树鼩KLF家族因子鉴定出来,并对基因家族全长以及锌指结构域进行系统分析。相关成果在线发表在杂志Oncotarget上,昆明动物所、昆明理工大学联合培养硕士研究生邵明、葛广哲及昆明理工大学硕士研究生刘文婧为论文的共同第一作者。
树鼩作为与人类亲缘关系较近的类灵长类动物,因具有个体较小、繁殖快、易于饲养等传统动物模型的优点,正在成为一种新型而有潜力的疾病动物模型。昆明动物所在2013年解析出树鼩基因组以后,近年来陆续建立了树鼩乳腺癌、非酒精性脂肪肝、2型糖尿病、疱疹病毒感染的脑炎和肌膜炎、抑郁症等疾病模型。
KLF基因家族是一类含有三个高度保守的串联锌指结构的转录因子家族,KLF因子调控基因的表达,在动物细胞的增殖、分化、衰老、死亡以及癌变中起着重要作用。前期研究发现KLF5在基底型乳腺癌干细胞维持中起着关键的作用。KLF家族在许多物种中得到鉴定,包括人类、小鼠、猪、鸡、斑马鱼、线虫等,但树鼩KLF基因家族的系统发生及表达谱还没有研究报道。
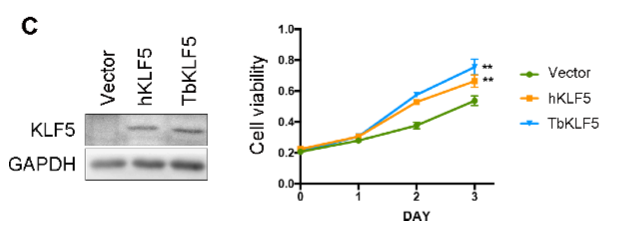
肿瘤生物学学科组研究发现,通过氨基酸全长进化树分析,树鼩17种KLF因子在物种间保守。树鼩高度保守的锌指结构与人有超过平均97%的同源性,略高于小鼠。锌指结构进化树分析发现,树鼩KLF1,2,5,17在进化地位上明显比小鼠更接近于人。对高度可变的N端功能基序的分析表明,树鼩KLF3,8,12有保守的CtBP结合序列PVDLS/T,树鼩KLF9,10,11包含保守的Sin3A结合序列AA/VXXL。核定位(NLS)序列比对发现,树鼩KLF1,4,8,13在N端拥有高度保守的NLS序列。通过RT-qPCR和WB揭示了树鼩KLF因子在不同组织中不同的表达模式。最后对树鼩KLF5序列单独分析发现,KLF5蛋白序列中存在重要的翻译后修饰位点,比如S153磷酸化位点,K369乙酰化位点,K162和K209 类泛素化(SUMO)化位点都在树鼩保守存在。泛素连接酶Fbw7以及WWP1识别的KLF5蛋白CPD和PY序列也同样严格保守,且树鼩KLF5与人类KLF5都具有促进细胞增殖的功能。该研究首次对树鼩KLF基因家族进行鉴定及分析,发现树鼩KLF基因家族在进化上较保守,比小鼠更接近于人。树鼩KLF蛋白结构与功能和人体中同源家族基因高度类似。研究结果为进一步以树鼩为疾病动物模型研究KLF家族因子与疾病的关系奠定了基础。
该研究得到国家自然科学基金和中科院昆明动物所一三五项目资助。


昆明动物所对树鼩KLF基因家族全长及锌指结构进行系统分析
