昆明植物所在托品烷生物碱生物合成途径的进化起源方面取得重要进展
在植物进化的特定时期,植物获得了生产结构丰富且功能多样的天然产物的能力,从而呈现出结构相似的天然产物集中于系统发育树上近源物种的分布特征;但部分结构相似的天然产物却零散地分布于系统发育树上远源物种中,如托品烷生物碱、类固醇和环烯醚萜等。现在,科学家们主要是通过单个酶的独立进化来阐明天然产物零散的分布特征。实际上,天然产物的生物合成涉及一系列酶的协同参与;然而,这一系列酶是如何演变的却知之甚少。基因复制及后续的新功能化作为植物天然产物结构多样性的主要贡献者,故通过基因组分析来阐明天然产物生物合成基因的复制事件,结合利用蛋白结构分析来揭示酶新功能化产生的关键氨基酸,可揭示天然产物生物合成酶的进化起源和演变历程。
托品烷生物碱(TAs)是一类由吡咯环和哌啶环骈合为托品烷基本骨架的生物碱,零散地分布于不同的科属植物中,如茄科、古柯科和旋花科等。其中,茄科来源莨菪碱和古柯科来源可卡因作为使用历史悠久的天然药物,现今仍在临床上广泛使用。中国科学院昆明植物研究所植物化学与西部植物资源持续利用国家重点实验室,黄胜雄研究员带领的天然产物生物合成及药用植物资源攻关团队长期聚焦于托品烷生物碱的生物合成研究,取得了一系列重要突破,专题组解析了一系列莨菪碱和可卡因的生物合成酶,如参与托品烷骨架前体构建的聚酮合酶PYKS和PKS1/2(Nat Commun, 2019, 10, 4036; Nat Commun, 2022, 13, 4994)、负责莨菪碱酯化步骤的关键酶(New Phytol, 2020, 225, 1906)、催化莨菪醛生成莨菪碱的氧化还原酶HDH(ACS Catal, 2021, 11, 2912)、以及负责可卡因托品烷骨架形成的芽子酮合酶CYP81AN15和芽子酮甲基转移酶MT4(J Am Chem Soc, 2022, 144, 22000),基本实现莨菪碱和可卡因完整合成途径的解析。
在此基础上,黄胜雄专题组完成了产莨菪碱的三分三植物和产可卡因的古柯植物的高质量基因组的组装和注释,为研究托品烷生物碱生物合成酶的进化提供了可靠的遗传信息。随后,通过比较基因组和蛋白结构分析,阐明了莨菪碱和可卡因生物合成中5对具有相似功能的合成酶(PMT/SPMT, PYKS/PKS, CYP82M3/CYP81AN15, TRI/MecgoR和LS/CS)及古柯特有的芽子酮甲基转移酶(MT4)的独立进化(图1)。其中,古柯科SPMT是由SPDS通过古六倍体复制事件演变而来,不同于茄科PMT由SPDS通过串联复制事件演变而来(图1B、图1C);茄科CYP82M3和古柯科CYP81AN15采用不同的策略来将相同的底物MPOA氧化环化为相同的产物芽子酮(图1D),且CYP81AN15是由古老的串联复制基因通过新功能而来;古柯植物中负责可卡因2-羧甲基基团合成的芽子酮甲基转移酶MT4,是由水杨酸甲基转移酶MT1经古柯特有的复制事件以及关键的E216和S153位点突变而来(图1E)。因此,从基因组和结构生物学这两个层面,对茄科和古柯科植物中TAs整条生物合成路径化学趋同而基因独立进化的揭示,为植物天然产物的进化研究提供了一个经典的案例;同时,这一整条生物合成途径趋同进化的发现也提醒我们:在研究远源物种中结构相似的天然产物生物合成时,完全基于酶同源性或关键保守位点来发现具有相似功能的生物合成酶需要谨慎。
以上研究成果以Genomic and structural basis for evolution of tropane alkaloid biosynthesis为题,发表在美国科学院院刊PNAS上。黄胜雄专题组的王永江博士后、田恬博士研究生和余佳益硕士研究生为本论文的共同第一作者,黄建萍副研究员和黄胜雄研究员为通讯作者。以上研究工作得到了国家合成生物学重点研发计划、中科院战略性先导科技专项、国家自然科学基金、云南省“兴滇英才支持计划”等项目资助。
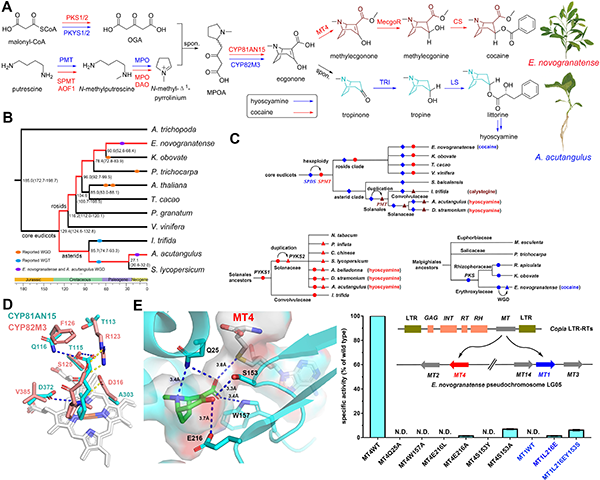
图1A: 莨菪碱和可卡因相似的生物合成路径;图1B: 三分三和古柯的系统发育树;图1C: SPMT、PMT、PYKS和PKS的进化历程;图1D: 芽子酮合成酶不同的催化策略;图1E: 芽子酮甲基转移酶的催化机制及演化
